近日,学院申涛课题组联合中国科学技术大学第一附属医院、燕山大学等单位,在 Nature旗下期刊《Communications Biology》(中科院一区 TOP)发表基因组超保守区域(UCRs)研究成果,系统更新 UCRs 注释体系并揭示其分类功能,为脑发育机制解析与脑疾病诊疗提供新靶点和潜在标志物。
UCRs 是在人、大鼠、小鼠基因组中序列完全保守的特殊元件(Bejerano,G.et al.Science,2004),其在脑发育与脑疾病中的调控机制长期不明。研究团队突破传统局限,整合34个跨物种最新参考基因组,对经典481个UCRs 重新注释验证,最终确立479个UCRs的更新目录。这些UCRs长度多集中在 200-400碱基对,具有低GC含量、低SNP密度但高有害突变比例的特征,其进化可追溯至鱼类,在脊椎动物中数量显著增加,提示这类元件可能与脊椎动物特有的器官发育密切相关,为后续功能研究锚定了核心方向。
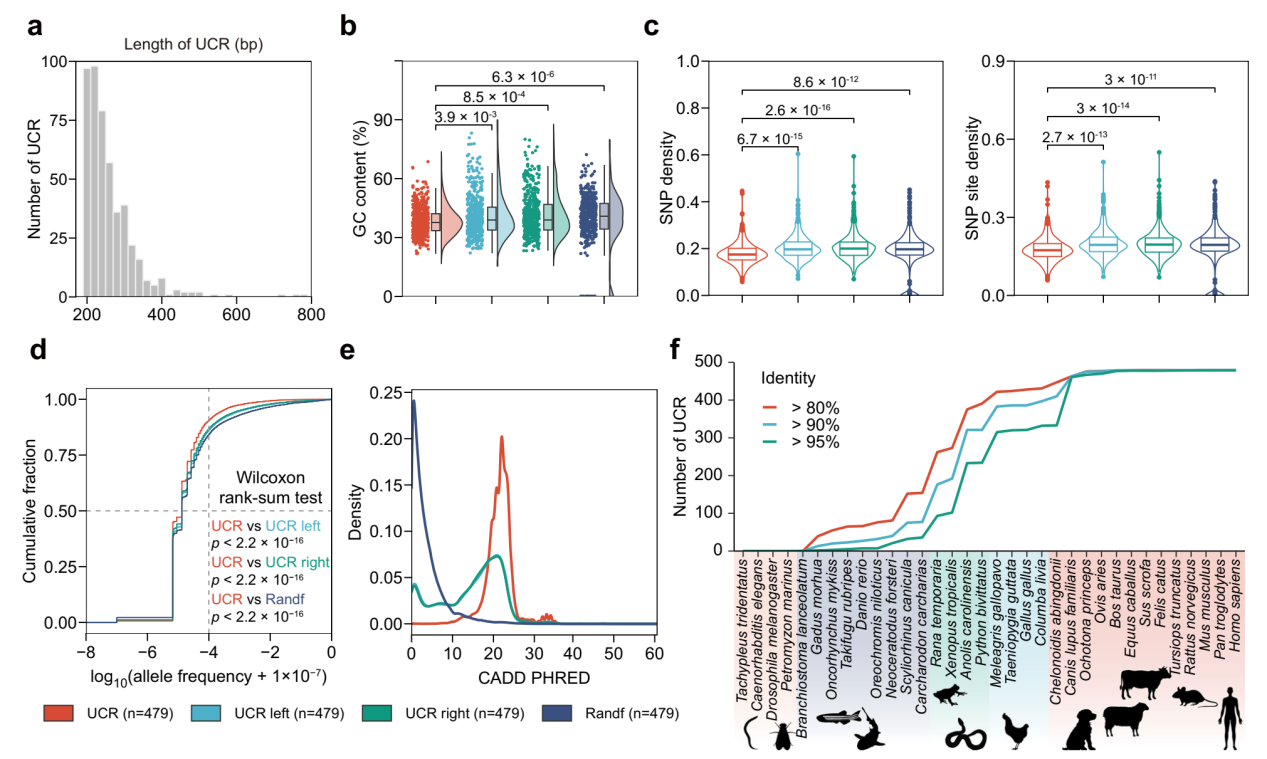
图1 UCR特征分析及进化溯源
基于基因组位置与功能,研究团队将 479个UCRs分为三类:TypeI位于蛋白编码基因内,通过调控剪接因子影响脑发育相关基因可变剪接;TypeⅡ与脑组织特异性长链非编码 RNA共定位,构建的诊断模型对低级别胶质瘤、胶质母细胞瘤识别度极高,部分关联RNA可用于预后评估;TypeⅢ位于基因间区,约46%与增强子样元件重叠,可调控脑发育关键基因。
该研究建立“UCRs 分类-功能-脑发育/疾病”关联框架,为神经发育障碍治疗和脑胶质瘤诊断提供新方向。研究得到国家自然科学基金、芜湖市科技计划项目和分子酶学与分子检测省级创新团队项目的支持,论文以“Characterizing and Decoding Ultraconserved Regions Uncovers Their Regulatory Significance in Human Brain Development and Disorders”为题发表,安徽师范大学为论文第一完成单位,安徽师范大学申涛课题组研究助理、中国科学技术大学附属第一医院及生命科学与医学部硕士毕业生柏运为该工作的第一作者,申涛为通讯作者,燕山大学汪梅婷副教授为共同通讯作者。研究还得到了中国科学技术大学附属第一医院汪香婷教授课题组的大力支持。
论文链接:https://www.nature.com/articles/s42003-025-09115-3

